Für das heutige Türchen habe ich aufgeschrieben, wie wir in “Modernes Publizieren” ein wiss. Journal in GitLab abbilden und Artikel in Pipelines produzieren. Dass wir mit diesem Ansatz effizient sind, konnten wir bei der Produktion der 37 Backlistartikel des Journals kommunikation@gesellschaft feststellen. Dort wir haben es erreicht, in einem Gang HTML- und PDF-Versionen aus einer Markdownquelle zu produzieren.
Für die Entwicklung der Journalstruktur in GitLab waren unsere Ziele
- einen möglichst hohen Grad von Automation und Reproduzierbarkeit für Nachnutzende unseres Ansatzes zu erreichen
- Updates, die sich aus der weiteren Entwicklung ergeben, möglichst effizient und fehlerarm einzuspielen
- Redundanzen möglichst zu vermeiden
Struktur
Die folgende Abbildung gibt am Beispiel des Journals kommunikation@gesellschaft wieder, wie wir unsere Journalstruktur in GitLab aufgebaut haben:
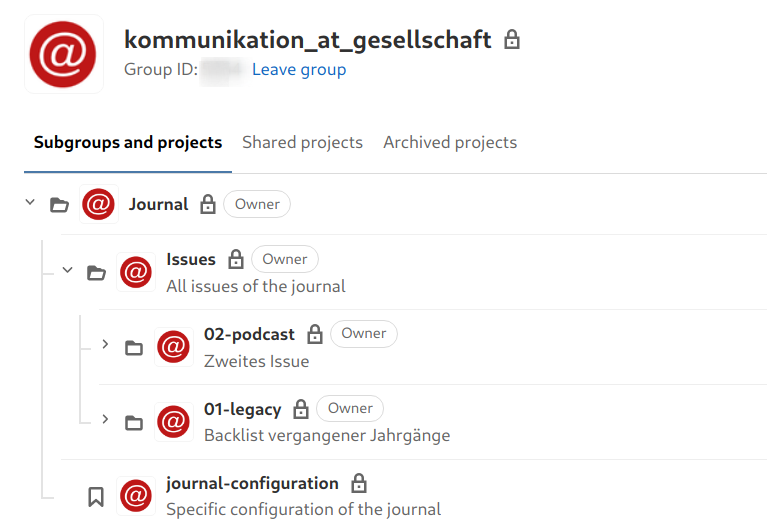
Journal-Struktur in GitLab
Wir haben für die gesamte Ordner- bzw. Gruppenstruktur in GitLab entschieden: “Alles ist ein Journal!". Das heißt, dass wir auch nur für einen einzigen Artikel immer gleich eine Journalstruktur einrichten, die entsprechend skaliert, wenn ein zweiter oder dritter oder auch nur ein Dummy-Artikel zum Ausprobieren geschrieben wird.
Das folgende Skript legt diese Struktur auf einem Client an und ist ganz nützlich, wenn die entsprechenden Kenntnisse für die Arbeit mit pandoc, LaTeX und make vorhanden sind. Wir setzen aber gerade deshalb GitLab ein, weil wir wissen, dass viele Interessierte gar nicht über einen Rechner mit Adminrechten verfügen und daher auch die notwendige Software nicht installieren können.
#!/bin/bash
NAME=${1:-scholarly-journal}
# Create the journal folder
mkdir $NAME
# Create the journal configuration
cd $NAME/
git clone https://collaborating.tuhh.de/hos/modernes-publizieren/offen/software/konfigurationen/journal-configuration.git
# Create pandoc-scholar
git clone https://github.com/pandoc-scholar/pandoc-scholar
# Create the pandoc templates
git clone https://collaborating.tuhh.de/hos/modernes-publizieren/offen/software/templates/pandoc-templates.git
# Create an issue folder
mkdir Issues
# Create two issue folders
cd Issues
mkdir 01-issue
mkdir 02-issue
# Create an example article in first issue
cd 01-issue
git clone git@collaborating.tuhh.de:hos/modernes-publizieren/offen/software/scaffolds/scholarly-article.git an-article
cat << EOF
---------------------------------
Done!
You can now build the example article:
$ cd ${NAME}/Issues/01-issue/an-article
$ make all
----------------------------------
EOF
Wie im Skript zu sehen, werden einige Ordner angelegt und dann eine globale Journalkonfiguration, die pandoc-Templates für HTML, PDF (und bald auch JATS!) sowie die aktuelle Version von pandoc-scholar geklont. Wir haben alles in diese Konfiguration gelegt, um mit make all einen Dummyartikel generieren zu können.1
Die analoge Struktur dazu haben wir auch in GitLab abgebildet und die Pfade dort entsprechend den lokalen Pfaden angelegt, damit eine nahtlose Produktion lokal und remote möglich ist. Heißt konkret: Artikel, die auf dem Client in der gezeigten Struktur geschrieben werden, laufen auch reibungslos in GitLab durch.
Dafür muss die oben gezeigte und im Folgenden noch einmal expandiert abgebildete Struktur vorhanden sein:

Artikel in der Journal-Struktur
Das heißt: Es gibt eine Gruppe Journal in GitLab, die eine Gruppe Issues enthält, die wiederum Gruppen für die einzelnen Ausgaben enthält. Jedes Issue enthält dann die wiss. Artikel, und zwar für jeden ein eigenes GitLab-Projekt.
Konfigurationen
Außerdem gibt es ein Projekt auf der Ebene von Journal, das die Konfiguration des Journals beinhaltet und von der erwartet wird, dass sie sich nicht ändert bzw. dezidiert überschrieben wird (vgl. journal-configuration, erste Abb.). Hier stehen bspw. Angaben zu den verwendeten Schriften oder pandoc-Variablen.
---
documentclass: scrartcl
classoption:
- abstract=on
- headlines=5
- headheight=5cm
lang: de-de
fontsize: 9pt
geometry:
- top=3.5cm
- left=2cm
- right=2cm
- bottom=2.8cm
sansfont: "OpenSans"
sansfontoptions:
- Path=<?SWAPFIRE_PATH?>/journal-configuration/fonts/
- UprightFont=*-Regular,
- BoldFont=*-SemiBold
- ItalicFont=*-Italic
- Scale=1
mainfont: "Merriweather"
mainfontoptions:
- Path=<?SWAPFIRE_PATH?>/journal-configuration/fonts/
- UprightFont=*-Regular,
- BoldFont=*-Bold
- ItalicFont=*-Italic
- Scale=1
linestretch: 1.3
links-as-notes: false
colorlinks: true
papersize: a4paper
reference-section-title: Referenzen
linkReferences: true
figPrefix: "Abb."
figureTitle: "Abbildung"
tblPrefix: "Tab."
tableTitle: "Tabelle"
[...]
---
Außerdem haben wir die Konfiguration für pandoc-scholar in diesem Projekt abgelegt und können hier bspw. auch sagen, welche Ausgabeformate generiert werden sollen:
# Name of the input file
ARTICLE_FILE = draft.md
# Path and prefix of output files
OUTFILE_PREFIX ?= build/outfile
# Main JSON file; contains article plus enriched metadata
JSON_FILE ?= $(OUTFILE_PREFIX).enriched.json
# Default extensions to be generated
DEFAULT_EXTENSIONS ?= html pdf slim.html
[...]
GitLab erlaubt es, CI/CD-Konfigurationen in übergeordneten Gruppen zu bündeln, sodass sie für alle darunterliegenden Gruppen und Projekte gelten. Für unser Journal machen wir uns das zunutze, indem wir sogenannte Runner Variables so hoch wie möglich in der Gruppenhierarchie definieren, um sie nicht nur für ein Journal, sondern gleich für mehrere nutzen zu können. Hierbei hatten wir den Verlagskontext im Kopf, wo ggf. zahlreiche Journals in Produktion sind.
Weitere Konfigurationen, die speziell für ein Journal, ein Issue oder einen Beitrag gelten, haben wir in den CI/CD-Settings weiter unten, also bis auf Projektebene, gespeichert.
Pipelines in GitLab werden in der Datei .gitlab-ci.yml konfiguriert. Dies geschieht bei uns in verschiedenen Stages, sodass wir das erzeugte HTML bspw. auch auf einen Webserver deployen können, um dort mit Hypothesis Reviewprozesse zu ermöglichen.
[...]
build:
stage: build
script:
- mkdir -p ~/.ssh
- chmod 700 ~/.ssh
- apk add git make ttf-linux-libertine
# Rebuild font cache
- fc-cache -fv
# Install necessary LaTeX packages
- tlmgr install makecell multirow floatflt footmisc textpos changepage latexmk
# Create journal structure
- mkdir -p Issues/issue/article
# Move all files from root level in the repo to the new structure
- ls -1A | grep -v ^Issues | xargs -I{} mv {} Issues/issue/article
# Create the target folder for the artifacts to be deployed
- mkdir -p build
# Clone the general journal configuration
- git clone https://gitlab-ci-token:${CI_JOB_TOKEN}@collaborating.tuhh.de/hos/modernes-publizieren/pre-submission-stage/kommunikation_at_gesellschaft/journal/journal-configuration.git
# Clone current version of pandoc-scholar
- git clone https://github.com/pandoc-scholar/pandoc-scholar
# Clone the general templates
- git clone https://collaborating.tuhh.de/hos/modernes-publizieren/offen/software/templates/pandoc-templates.git
- cd Issues/issue/article
# Create another build folder for the make targets
- mkdir -p build
[...]
Wie im Skript zu sehen ist, werden zur Laufzeit verschiedene Repos aus GitLab und von GitHub dazugeklont, um immer “frisch” zu produzieren. Dieser Ansatz birgt natürlich die Gefahr, dass die Pipeline wegen Inkompatibilitäten bricht, zwingt aber auch dazu, stets kompatibel zu den Updates zu bleiben. Diese Arbeit ist zeitlich nicht zu unterschätzen.
Zwischenfazit
Die bisherigen Abschnitte sollen Folgendes verdeutlichen: Analog zur Softwareentwicklung triggern Änderungen in den Artikeln (Projektebene) die zugehörige Pipelines. Beim Lauf der Pipeline werden Templates, Journalkonfiguration und pandoc-scholar als Aufsatz von pandoc dazugeklont. LaTeX steckt schon im Docker-Image, die Ordner werden dynamisch bei jedem Durchlauf angelegt. Damit haben wir ein System, in dem wiss. Artikel analog zu den gängigen Praktiken der Softwareentwicklung (kollaborativ) produziert werden können.
Projektkonfiguration
Zuletzt gilt es noch, einen Blick auf Struktur und Konfiguration der Artikel zu werfen. Hierbei handelt es sich um GitLab-Projekte, für die gilt: Markdown ist der “Quellcode”, HTML und PDF sind die “Zielartefakte”, die von der Pipeline produziert werden.
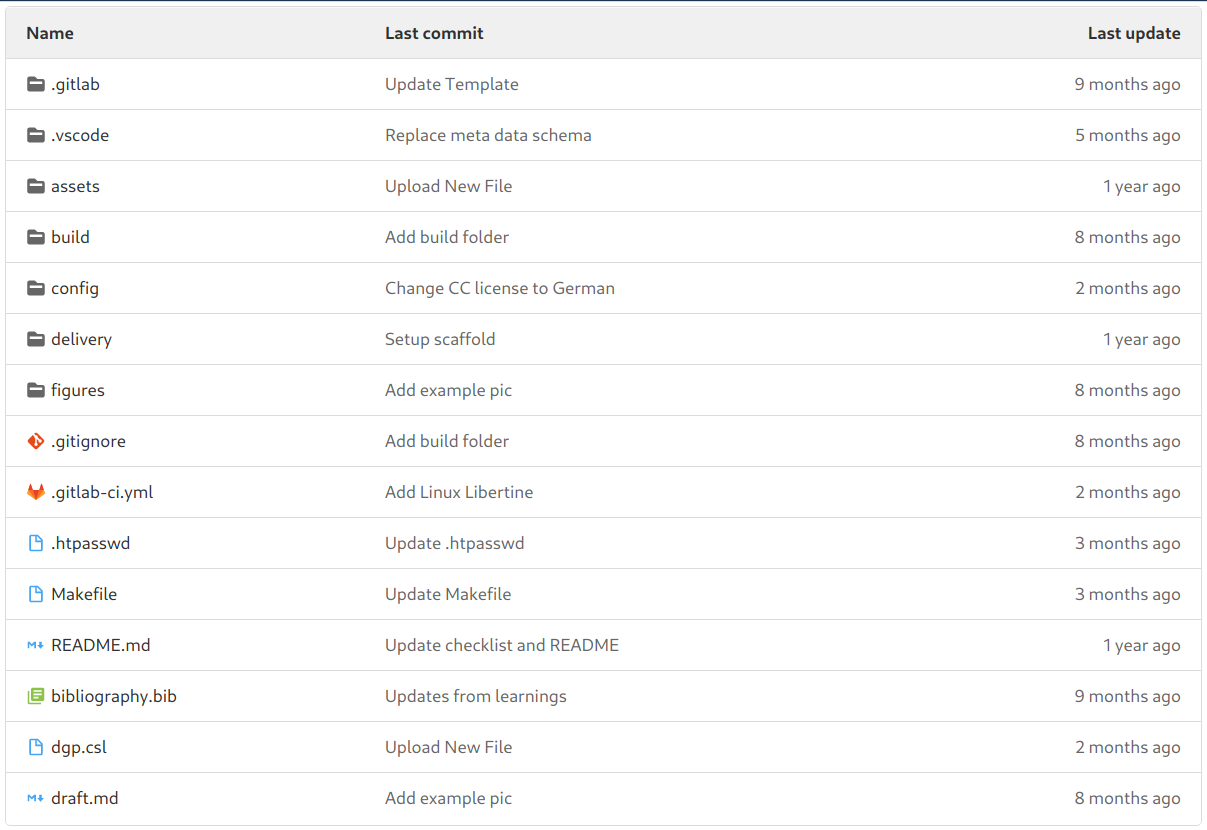
Die wichtigsten Ordner und Dateien im Überblick:
- build
- Zielordner für Artefakte; hier ausnahmsweise mit eingecheckt, um Fehler in der Produktion zu vermeiden (historisch gewachsen…)
- config
- enthält die Datei
article-metadata.inc.yml, die alle artikelspezifischen Metadaten speichert (s.u.) - delivery
- enthält alle angelieferten Dateien, also bspw. Word-Dokument, Bibtex-Datei und/oder Abbildungen
- figures
- enthält die Abbildungen des Beitrags
- dgp.cls
- Zitationsstil des Journals (könnte auch auf Journalebene wandern)
- draft.md
- der eigentliche Text des Artikels
Das folgende Snippet zeigt die kompletten Dummymetadaten eines Scholarly articles. Diese Struktur ist auch im Projekt gewachsen und von keinem Standard abgeleitet. Vielmehr sind wir den formalen Vorgaben des Templateentwurfs gefolgt und haben darauf geachtet, dass die Struktur im Netlify-Backend abgebildet werden kann (Türchen folgen!).2
---
copyright-holder: Klaus Honsky et al.
license:
- type: CC-BY
license-text: Dieses Werk steht unter einer Lizenz
Creative-Commons-Namensnennung 4.0 (CC-BY 4.0) International
license-url: https://creativecommons.org/licenses/by/4.0/deed.de
bibliography: ./bibliography.bib
title: |
Schreiben und publizieren unter Berücksichtigung der Werte von Open Science
subtitle: |
Ein Vorschlag
keywords:
- keyword: Lorem
- keyword: Ipsum
- keyword: Dolor
- keyword: Sit
- keyword: Amet
- keyword: Consetetur
author:
- name: Babette Petersen
institute:
- "2"
email: babette@petersen.de
orcid: 0000-0000-0000-0000
correspondence: false
- name: Klaus Honsky
institute:
- "1"
- "2"
email: klaus@honsky.de
orcid: 0000-00000-0000-0000
correspondence: true
page:
lang: de-DE
heading:
access: Open Access
label: Publiziert am
doi: "10.15460/modpub/2020.1"
footer:
publisher: Publiziert von Hamburg University Press
publisher-url: https://blogs.sub.uni-hamburg.de/hup/
date: Mai 2020 | Bd. 19 | Artikel 9
institute:
- id: 1
name: Staats- und Universitätsbibliothek Hamburg
- id: 2
name: Universitätsbibliothek der Technischen Universität Hamburg
link-citations: true
recommended-citation: "Honsky K, Petersen B (2020). Schreiben und Publizieren unter Berücksichtigung der Werte von Open Science. ModPub, 1, 1. doi: 10.15460/modpub/2020.1"
languages:
- id: de-DE
name: Deutsch
- id: en-GB
name: Englisch
csl: ./dgp.csl
quality:
reviewed: true
received: 2020-06-01T22:00:00.000Z
accepted: 2020-06-08T22:00:00.000Z
published: 2020-06-22T22:00:00.000Z
reviewers:
- name: Klaus Koslowski
orcid: "0000-0000-0000-0000"
institution: SUB
country: Deutschland
- name: Helga Hansen
orcid: "0000-0000-0000-0000"
institution: TUHH
country: Deutschland
contribution: Babette Petersen hat die Daten erhoben und den Beitrag verfasst.
data-loc: Alle relevanten Daten befinden sich innerhalb der Veröffentlichung.
financing: Diese Arbeit wurde finanziert durch verschiedene Geldgeber.
transparency: Die Autor*innen erklären, dass ihre Forschung ohne kommerzielle oder finanzielle Beziehungen durchgeführt wurde, die als potentielle Interessenskonflikte ausgelegt werden können.
---
Fazit
Mit der gezeigten Struktur und Konfiguration haben wir einen lauffähigen Single-Source-Publishing-Ansatz entwickelt, den wir produktiv nutzen. Gegenwärtig arbeiten wir daran, die Laufzeit der Pipelines zu verkürzen, indem wir noch mehr Konfigurationen und Abhängigkeiten in einem neuen Docker-Image kapseln. Damit ist es dann auch möglich, Beiträge in einem Journallayout lokal mit Docker zu produzieren - unabhängig von GitLab.
Die Repos, aus denen die vorgestellten Snippets stammen, sind alle öffentlich und warten darauf, ausprobiert zu werden!
- journal-setup.sh zum Anlegen einer Journalstruktur auf dem eigenen Rechner
- journal-configuration, globale Konfiguration für ein Journal
- Scholarly article, Beispielartikel, darin die Metadaten sowie eine CI/CD-Konfiguration